2 min to read
Chip-seq分析
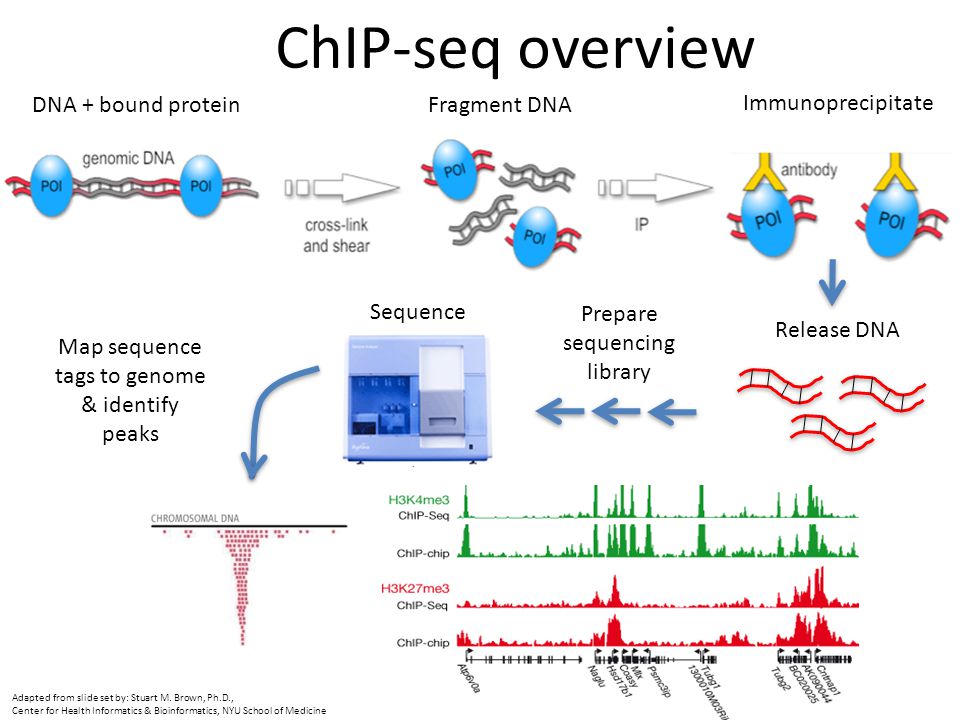
简介
ChIP是指染色质免疫沉淀,它通特异结合抗体将DNA结合蛋白免疫沉淀,可以用于捕获蛋白质(如转录因子,组蛋白修饰)的DNA靶点。
DNA和蛋白质交联(cross-linking),超声(sonication)将染色体随机切割,利用抗原抗体的特异性识别(IP),把目标蛋白相结合的DNA片段沉淀下来,反交联释放DNA片段,最后是测序(sequencing)。
motif是比较有特征的短序列。
实操
所需数据
ChiP-seq分析练习所需数据已上传至百度网盘, 提取码为sjey,欢迎下载。
所需工具
- linux
- bowtie
- samtools
- macs
- R语言
- ChIPseeker
- 在线网站
比对
#建立索引,为了减少占用的计算资源
bowtie-build bowtie_index/mm10.fa bowtie_index/mm10
#查看生成的结果,共6个文件
ls -l bowtie_index/*.ebwt
#结果
-rw-r--r-- 1 y570pc y570pc 59026331 10月 25 19:57 bowtie_index/mm10.1.ebwt
-rw-r--r-- 1 y570pc y570pc 23988656 10月 25 19:57 bowtie_index/mm10.2.ebwt
-rw-r--r-- 1 y570pc y570pc 458 10月 25 19:55 bowtie_index/mm10.3.ebwt
-rw-r--r-- 1 y570pc y570pc 47977298 10月 25 19:55 bowtie_index/mm10.4.ebwt
-rw-r--r-- 1 y570pc y570pc 59026331 10月 25 19:59 bowtie_index/mm10.rev.1.ebwt
-rw-r--r-- 1 y570pc y570pc 23988656 10月 25 19:59 bowtie_index/mm10.rev.2.ebwt
#比对
bowtie -m 1 -S bowtie_index/mm10 Oct4.fastq > Oct4.sam #'-S'表示输出格式为sam,'-m -1'表示reads多次map到参考序列时bowtie仅报道一次。
#查看结果
head -n 10 Oct4.sam
#部分结果
@HD VN:1.0 SO:unsorted
@SQ SN:chr1 LN:195471971
@PG ID:Bowtie VN:1.2.2 CL:"/usr/bin/bowtie-align-s --wrapper basic-0 -m 1 -S bowtie_index/mm10 Oct4.fastq"
SRR002012.45 0 chr1 125556548 255 26M * 0 0GACAGTGATTCATTATGCTGGAATGT IIIIIIIIIIIIBIIIIIIII7III$ XA:i:2 MD:Z:2A22A0 NM:i:2 XM:i:2
SRR002012.48 16 chr1 118910985 255 26M * 0 0CCGCAAAGGCCGAAGGAGGCGCAATC IIIIIII"III+IIFIIIIIIIIIII XA:i:1 MD:Z:11T14 NM:i:1 XM:i:2
SRR002012.187 16 chr1 130368773 255 26M * 0 0GACGGTCCATTACCCCAACTGCGTAC I-I*I&IIIIIAIIIIIIIIIIIIII XA:i:2 MD:Z:3T4C17 NM:i:2 XM:i:2
SRR002012.210 0 chr1 41092774 255 26M * 0 0ATCTGCACAAAGCTTAGAGTGGATAA I$III-IIIIIIIIIIIIIIII%III XA:i:2 MD:Z:1G3A20 NM:i:2 XM:i:2
SRR002012.240 0 chr1 21217509 255 26M * 0 0GCTCCAACTTTCCGACCCACAGAACA IIIIIIIIIIII/IIII)III.II5I XA:i:2 MD:Z:12A4A8 NM:i:2 XM:i:2
SRR002012.252 16 chr1 172408548 255 26M * 0 0TGAGAGATAATATCTCATGGGGGGGT F'IIIII=II+IIIIII;IIIIIIII XA:i:1 MD:Z:1C24 NM:i:1 XM:i:2
SRR002012.348 0 chr1 176643081 255 26M * 0 0GGCATGGGCTGACCACCTCGGGGTCT II(IIIII(I?I%IIIII1%&-*9I/ XA:i:2 MD:Z:2A9A13 NM:i:2 XM:i:2
#转sam文件为bam文件,bam为二进制文件,可以减少存储空间
samtools view -bSo Oct4.bam Oct4.sam
#排序
samtools sort Oct4.bam -o Oct4.sorted.bam
#对已排序的文件建立索引
samtools index Oct4.sorted.bam #h会生成索引文件Oct4.sorted.bam.bai
与Oct4.fastq文件处理类似,对gfp.fastq文件(对照组)也进行如上操作。
peak calling
macs14 -t Oct4.sorted.bam \
-c gfp.sorted.bam \
--format=BAM --name=Oct4 --gsize=138000000 \ #'--gsize'为比对的基因组大小
--tsize=26 --diag --wig #'--tsize'指定read大小
下游分析
peak注释
##安装依赖的包
options(repos="https://CRAN.R-project.org")
source("https://bioconductor.org/biocLite.R")
biocLite("TxDb.Mmusculus.UCSC.mm10.knownGene")
biocLite("ChIPseeker")
##设置工作路径
setwd("D://y570pc/yanshi")
##载入依赖的包
library("TxDb.Mmusculus.UCSC.mm10.knownGene")
library("ChIPseeker")
##载入数据
mm10<-TxDb.Mmusculus.UCSC.mm10.knownGene
macs<- readPeakFile("Oct4_peaks.bed")
##注释
anno <- annotatePeak(macs, TxDb=mm10)
##绘图
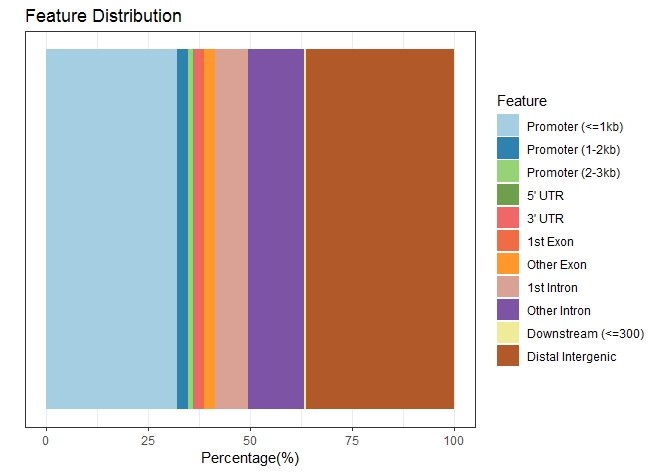
plotAnnoBar(anno)
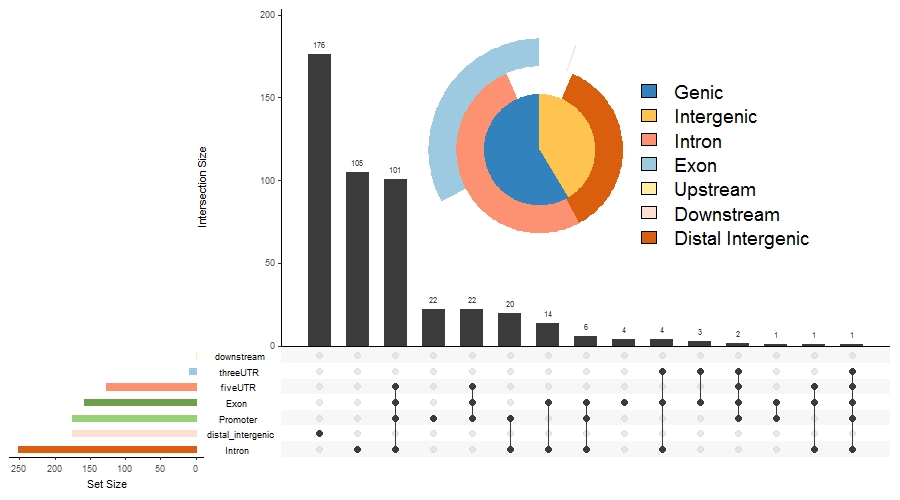
upsetplot(anno, vennpie=TRUE)
##保存数据
anno_df<-as.data.frame(anno)
write.csv(anno_df,"anno.csv")
结果如下:
motif分析
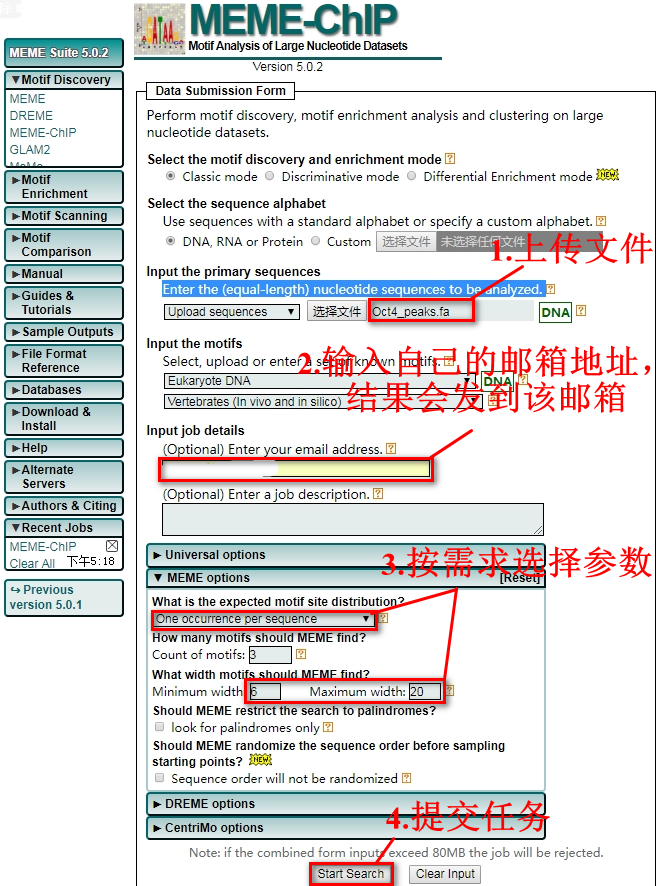
因为接下来分析用到的网站只支持fa格式的文件,需要将bed文件转化为fa文件。
bedtools getfasta -fi ./bowtie_index/mm10.fa -bed Oct4_peaks.bed -fo Oct4_peaks.fa
所需网站,按下图所示操作。
参考文献
[01]. ChIPseeker: an R package for ChIP peak Annotation, Comparison and Visualization
[02]. EBI: Next Generation Sequencing Practical Course
[03]. 学员分享-Chip-seq 实战分析流程
[04]. ChIP Seq Alignment and Processing Pipeline
[05]. NGS data analysis with R / Bioconductor: ChIP-Seq workflow
[06]. 一篇文章学会ChIP-seq分析(上)

Comments